10X Xenium Explorer1.2软件使用
Last updated on December 15, 2025 am
10X Xenium Explorer1.2软件使用
零、准备
分析前需要下载Xenium Explorer 软件,现在是1.2版本(2023.09)
官网下载地址:https://www.10xgenomics.com/support/software/xenium-explorer/downloads
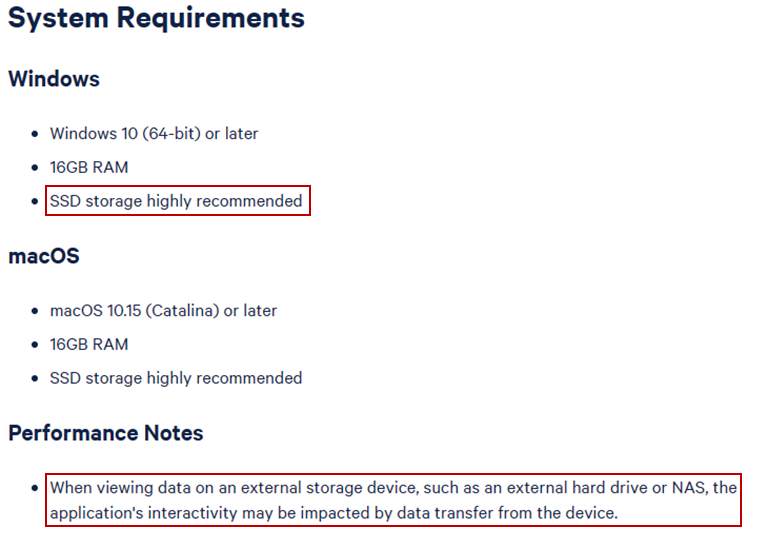
对电脑性能有要求,软件下载到固态硬盘所在的盘符(实验室Dell工作站C盘),勿放于外部硬盘或NAS
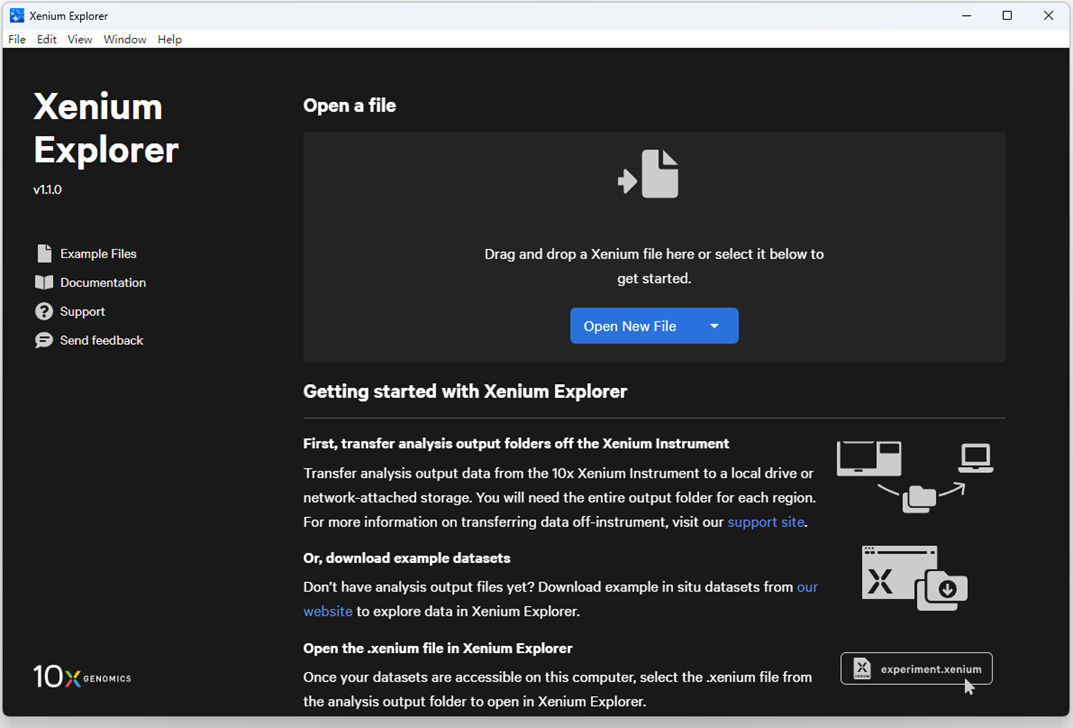
一、打开文件
在Xenium Explorer操作界面”Open New File”选择数据集,选择experiment.xenium打开
二、细胞注释
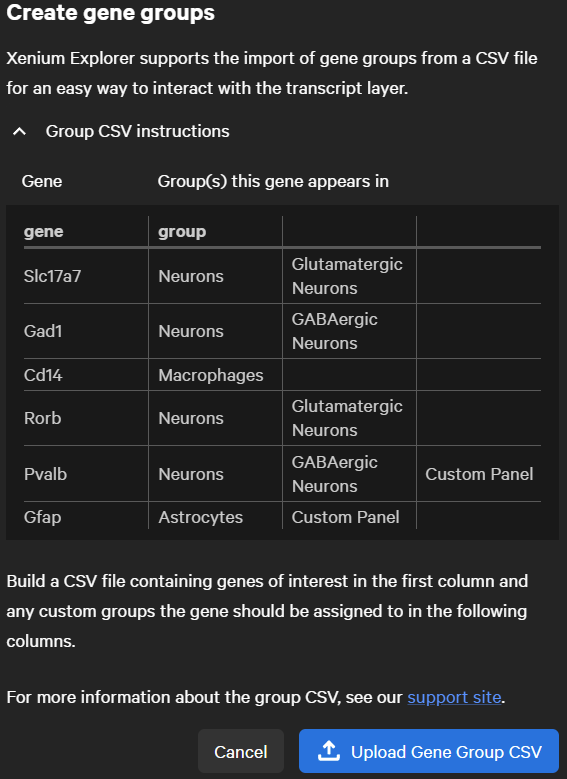
1.依据样式制做细胞注释csv文件
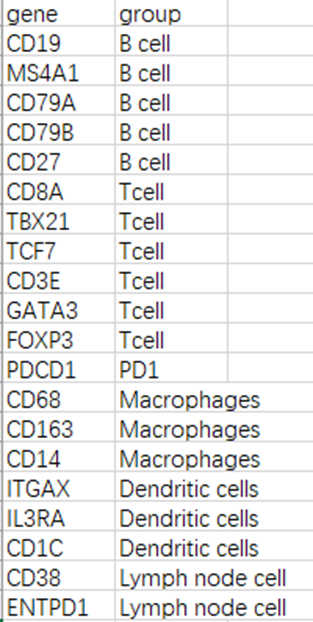
Xenium目前能检测到单个细胞上的380个基因,注释gene只能选择它提供的,注意Gene是否有别名。
以3946_1F文件为例,检测到的380个gene list在cell_feature_matrix文件夹下,将features.tsv.gz文件解压缩得到“features.tsv”文件
注:Xenium提供的这3个文件不是10X单细胞测序的标准文件,barcodes不是TCGA,不能用read10X读取。
3.Upload Gene Group CSV
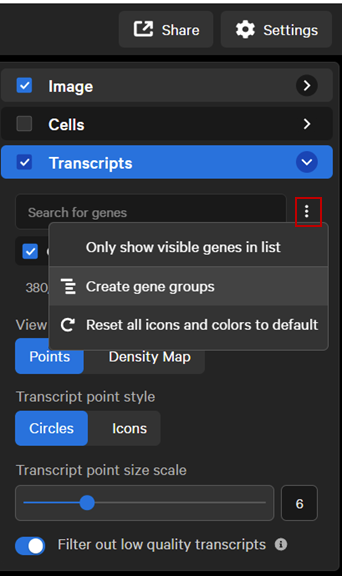
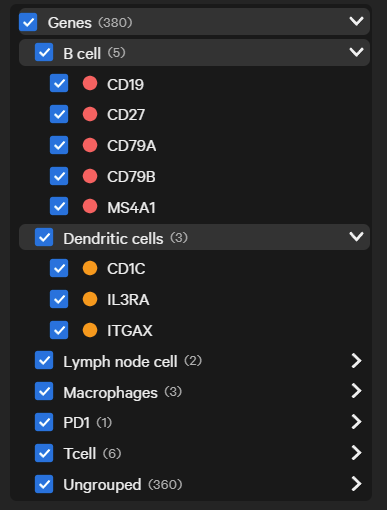
4.出现了我们自定义的细胞类型,按需更改颜色和图标;
三、文件信息
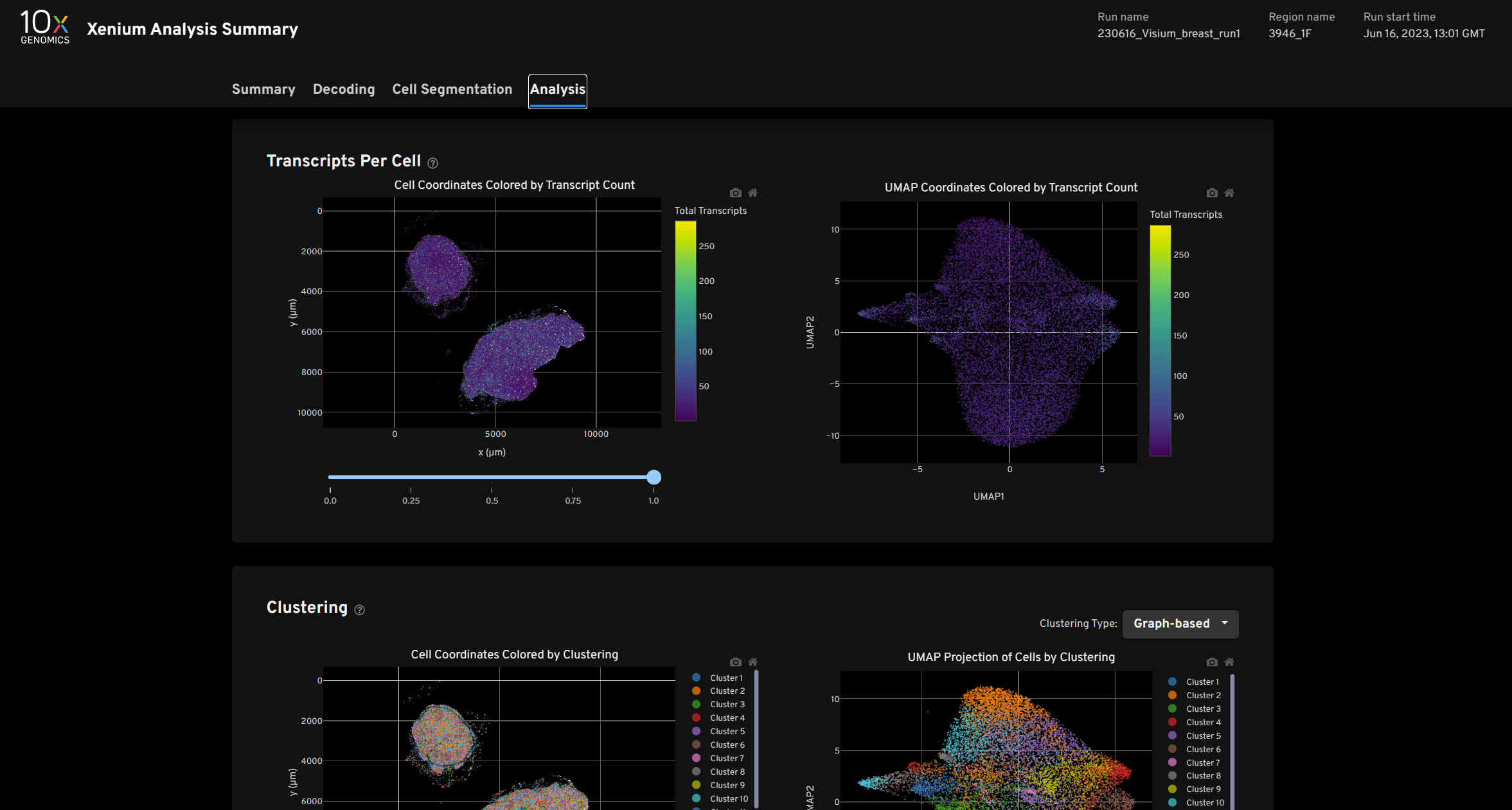
3946_1F文件夹下analysis_summary.html 提供了分析初步信息
四、H&E图像导入及对齐
H&E图像需要手动导入到Xenium explorer软件中,并对齐H&E图像与细胞位置图像。
https://www.10xgenomics.com/support/software/xenium-explorer/tutorials/xe-image-alignment
(1)H&E图像从转换为与Xenium Explorer兼容的格式
Xenium Explorer接受的图像文件格式要有.ome.tif后缀
这里是如何制作有.ome.tif后缀的图像文件:https://www.10xgenomics.com/support/software/xenium-explorer/tutorials/xe-image-file-conversion
需要条件
- QuPath 软件(已安装)
- Tifffile Python 库
(2)Image选择Add image,对齐图像
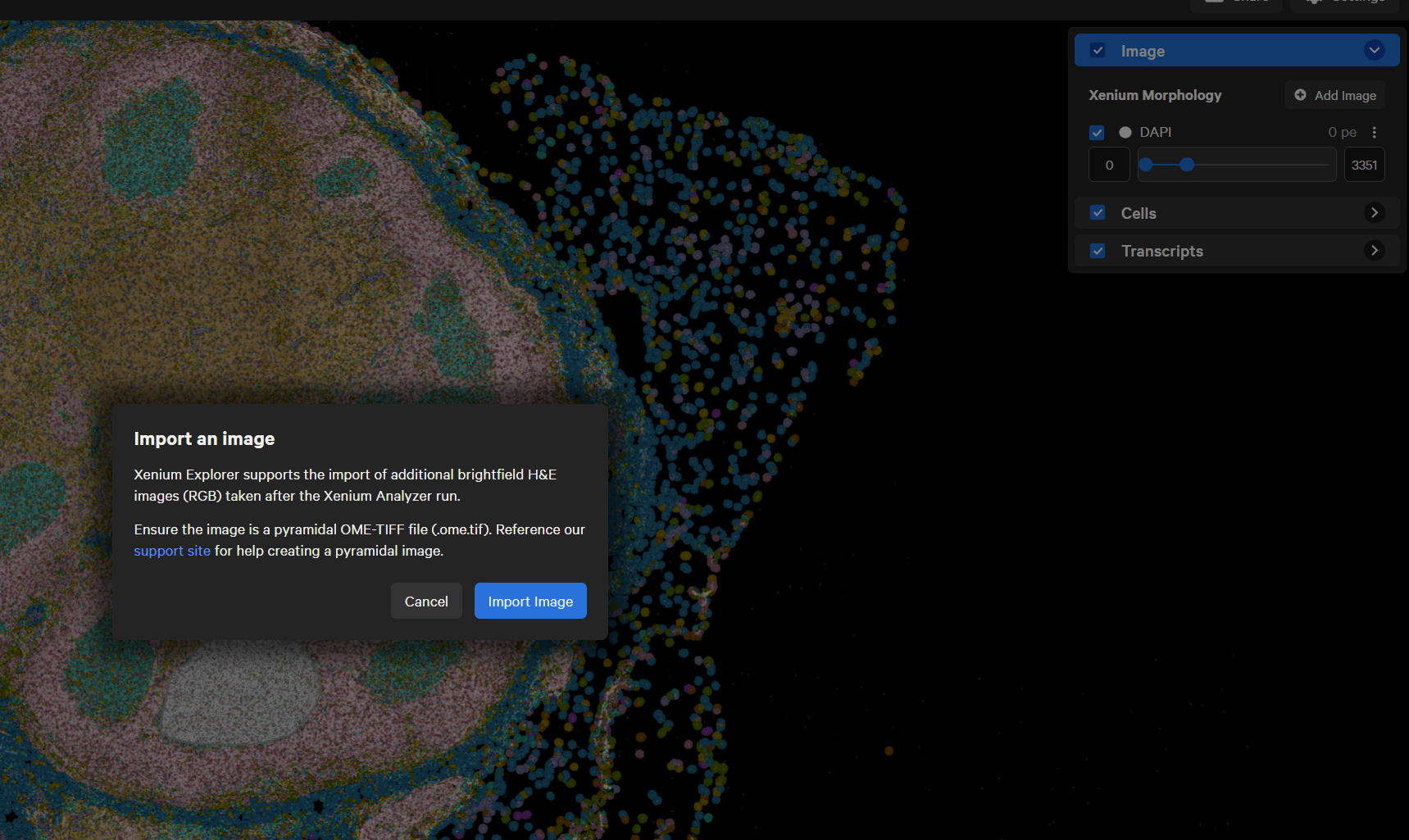
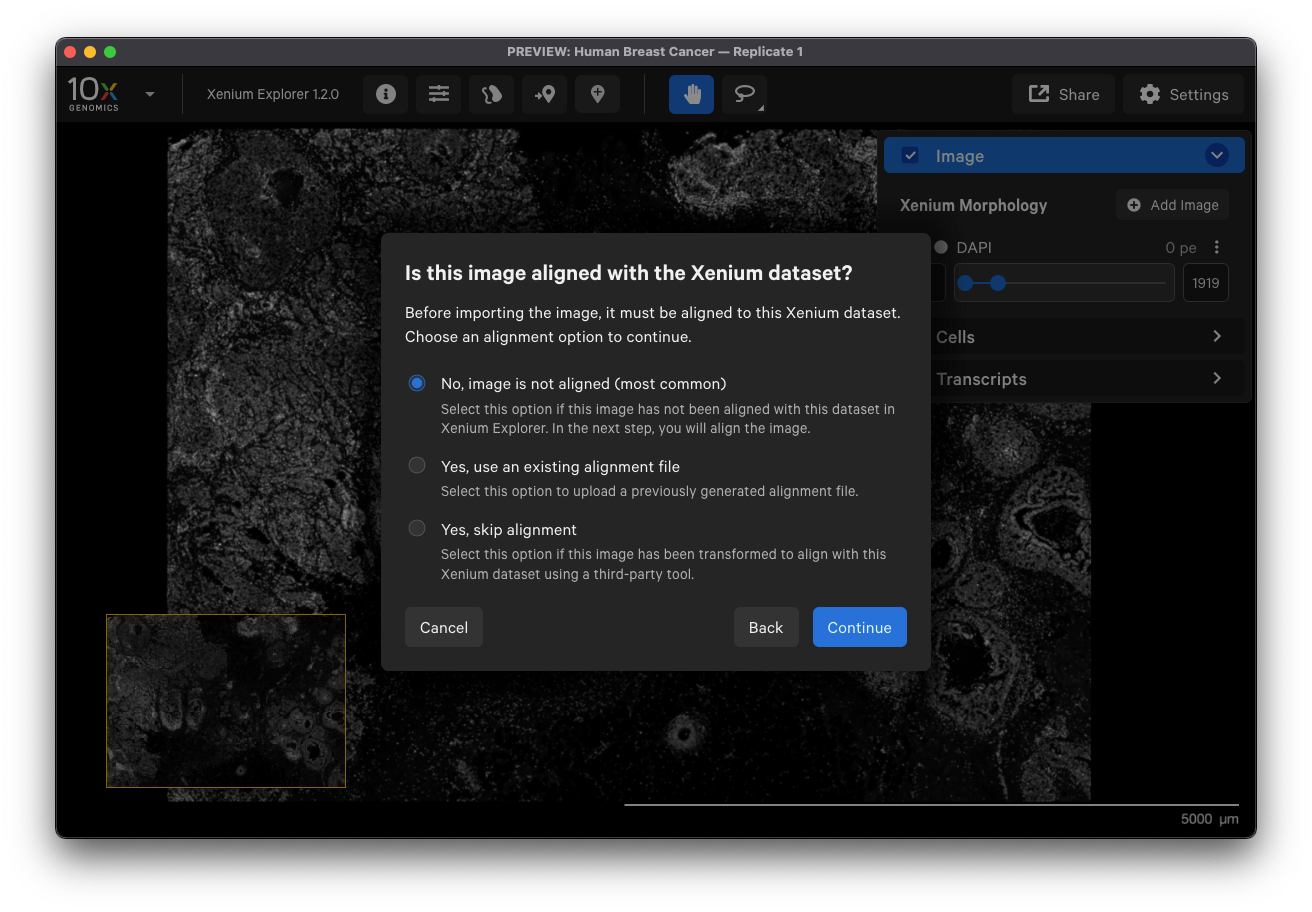
(3)选择图像是否对齐(推荐对齐)
三个选项,见官网教程。通过手动放置6个关键点就可辅助对齐,对齐后即导入成功。
https://www.10xgenomics.com/support/software/xenium-explorer/tutorials/xe-image-alignment
五、选区,下载Transcripts Data
1.手动选区,选择套索工具
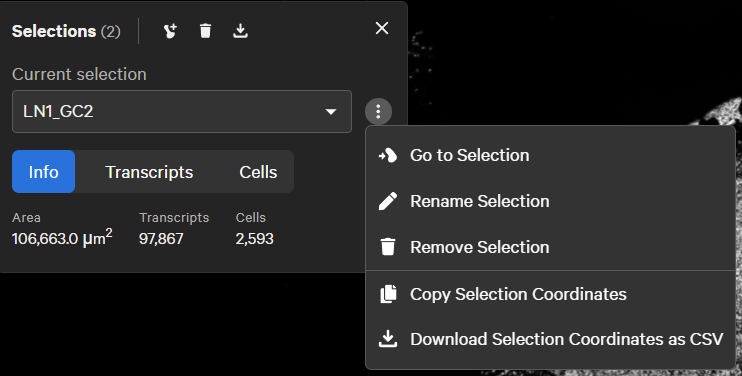
1.1选区重命名
2.界面右边栏Transcripts 全勾选,左边栏即可显示信息,点击Selection Transcript Stats 旁边的竖点下载。
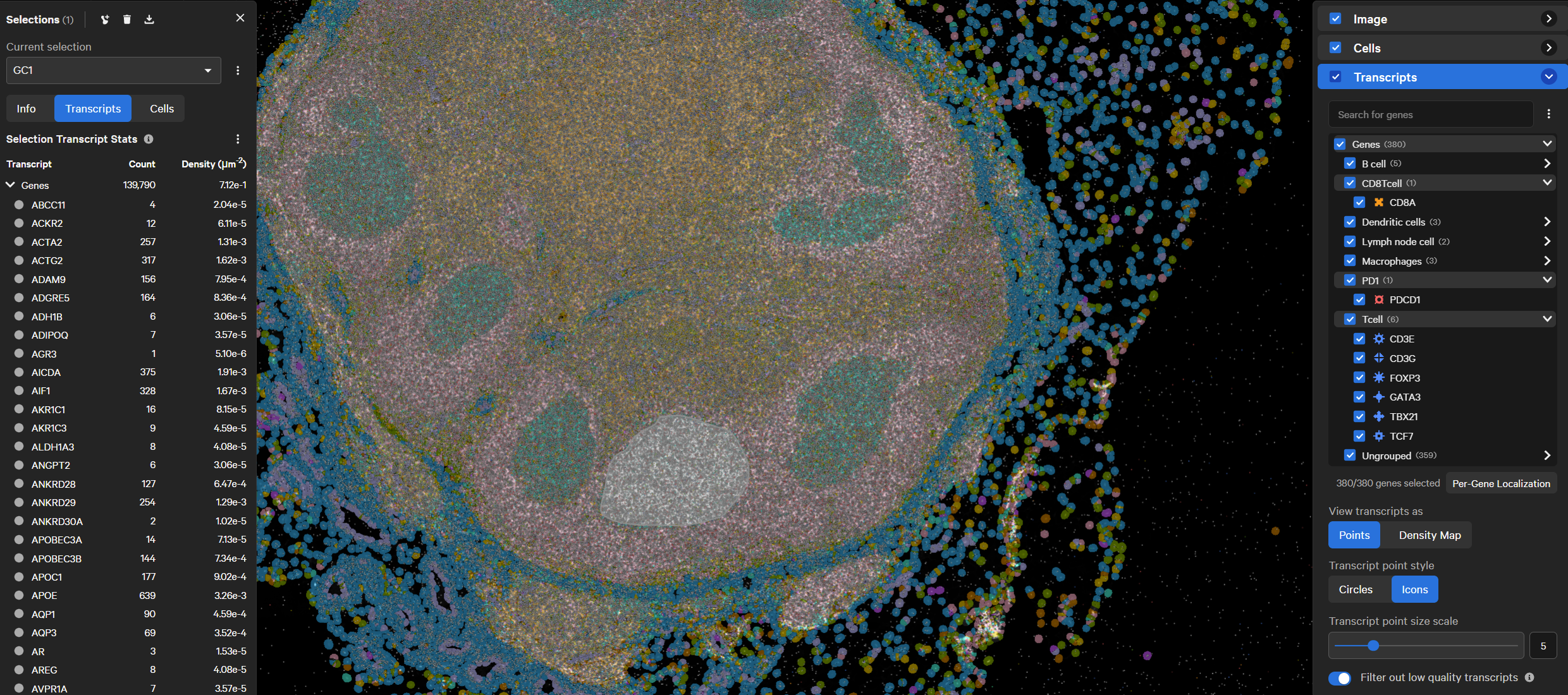
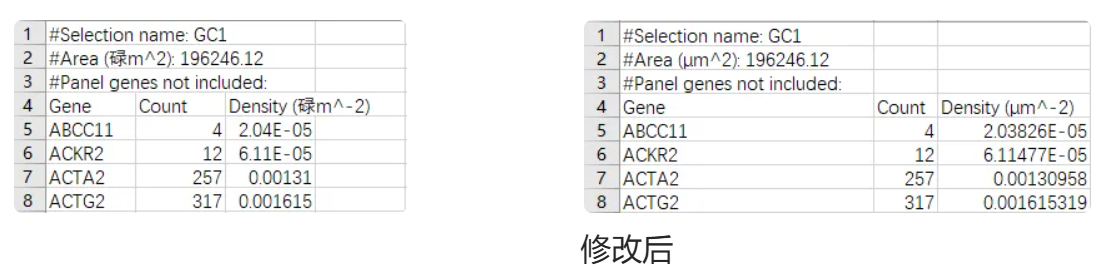
3.直接下载得到数据,其中µ显示成了碌,可能是保存为csv时分隔符号的问题。
解决办法:打开空白excel,选择“数据”——“自文本”,UTF-8编码,分隔符号选择逗号,保存为xlsx.
https://zhuanlan.zhihu.com/p/363126524
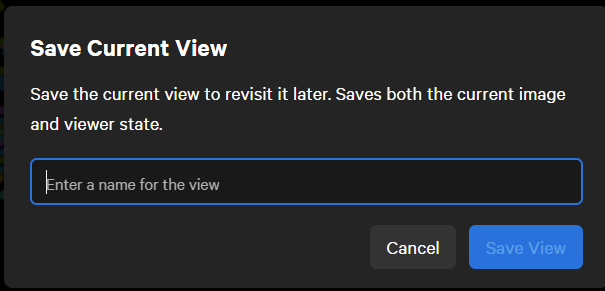
六、保存
save current view